DifPy - Diffusion on Graphs in Python [PL]
26 Apr 2020#python #difpy #multi-agent modelling #diffusion #social network analysis

W niniejszym artykule chciałbym przedstawić DifPy - pakiet do symulacji wieloagentowych napisany w języku Python, który jest obecnie na wczesnym etapie rozwoju. Główne funkcjonalności pakietu to tworzenie grafów do symulacji, przeprowadzanie symulacji wieloagentowych na grafach, wybór zestawu wierzchołków, które najlepiej rozprowadzają informacje po grafie, oraz badanie związku statystycznego cech przypisanych do węzłów, ze zdolnością tych węzłów do propagacji informacji. Kod źródłowy wraz ze wskazówkami dotyczącymi instalacji opublikowano w serwisie GitHub na licencji MIT - DifPy repository.
Pakiet DifPy składa się z 12 funkcji zawartych w 4 modułach - module inicjalizującym, symulacyjnym, optymalizacyjnym i modelująco-objaśniającym. Napisano pięć funkcji modułu inicjalizującego służących do tworzenia i modyfikowania grafów, obliczania ich podstawowych charakterystyk i wizualizacji. Moduł symulacyjny składa się z trzech funkcji, które umożliwiają przeprowadzanie symulacji wieloagentowych na grafach. Do obliczania zdolności węzłów do propagacji informacji wykorzystywana jest metoda Monte Carlo. Moduł optymalizacyjny jest odpowiedzialny za typowanie zadanego n najlepszych wierzchołków w danej sieci, które możliwie najlepiej rozpropagują informację. Zaprojektowano dwie funkcje – jedną wyznaczającą n wierzchołków na podstawie miary centralności – bliskości (ang. closeness), oraz drugą funkcję realizującą te samą funkcjonalność symulacyjnie za pomocą metody Monte Carlo i losowego przeszukiwania przestrzeni rozwiązań. W Module modelująco-objaśniającym zaimplementowano funkcjonalność identyfikacji cech jednostek w danej sieci społecznej, które najlepiej rozpropagują informację, lub wypromują produkt. Funkcjonalności tej odpowiadają dwie funkcje. Pierwsza z nich oblicza zdolność wierzchołków grafu do rozprowadzania informacji. Druga z nich wykorzystuje funkcję pierwszą – oblicza miary wierzchołków, zestawia je z cechami węzłów, tworzy model xgboost, a następnie oblicza istotność badanych cech.
Pakiet DifPy napisano z myślą o przeprowadzaniu eksperymentów na grafach reprezentujących rzeczywiste sieci społeczne. Prowadzenie eksperymentów symulacyjnych może wymagać odwzorowania danych o sieci, którymi dysponujemy, w graf biblioteki NetworkX. Artykuł o podstawach pracy z tą biblioteką można znaleźć tutaj - Social Network Analysis in Python - Introduction to NetworkX.
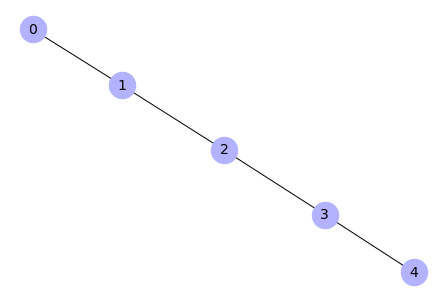
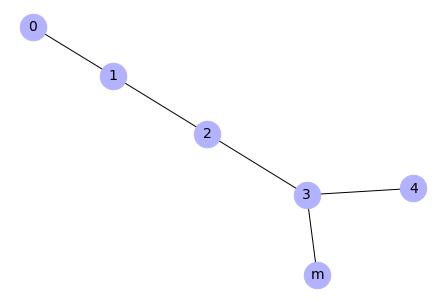
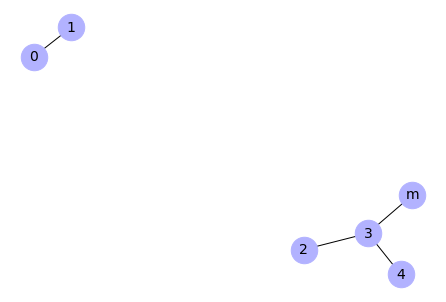
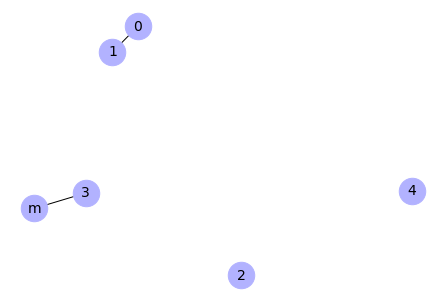
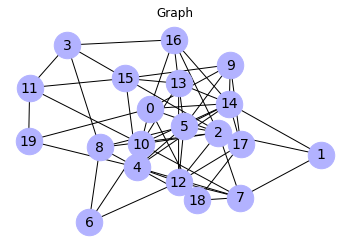
Rysunek 1. Struktura pakietu DifPy
Powyższy rysunek przedstawia strukturę pakietu DifPy rozpisaną na poszczególne moduły, funkcje i zależności tych funkcji. Korzeń drzewa reprezentuje pakiet DifPy. Węzły na pierwszym poziomie odzwierciedlają moduły pakietu. Węzły na trzecim poziomie oznaczają funkcje, natomiast czwarty poziom odnosi się do funkcji, które są wywoływane przez funkcje na trzecim stopniu drzewa.
Poniżej importowana jest biblioteka DifPy. Podczas uruchamiania jej funkcji automatycznie odnosi się ona do innych bibliotek takich jak NetworkX, czy Numpy, więc nie ma potrzeby ich dodatkowego importowania.
import difpy as dp
W dalszej części artykułu zaprezentowano działanie funkcji pakietu DifPy. Dane do przykładów generowane są w sposób losowy z wykorzystaniem modułu initialize.py, opierając się na mechanizmach losowego generowania sieci i cech zaczerpniętych z bibliotek NetworkX i Numpy. Ponadto do przetestowania funkcji z czwartego modułu badającej istotność cech, wygenerowano losowe zmienne z wykorzystaniem bibliotek Numpy, SciPy, math oraz statsmodels.
Contents:
1. Moduł inicjalizujący
2. Moduł symulacyjny
3. Moduł optymalizacyjny
4. Moduł modelująco-wyjaśniający
1. Moduł inicjalizujący
Moduł inicjalizujący initialize.py utworzono w celu ułatwienia generowania grafów i dostosowywania istniejących już grafów do przeprowadzenia symulacji.
Moduł inicjalizujący zawiera pięć funkcji. Funkcja graph_init() służy do utworzenia przykładowego grafu gotowego do przeprowadzenia symulacji. Funkcja draw_graph() jest funkcją pomocniczą i służy do wizualizacji grafu. Funkcja graph_stats() jest również funkcją pomocniczą, służy do obliczenia podstawowych statystyk grafu i wizualizacji sieci. Funkcja add_feature() odpowiada za dodawanie istniejących już wcześniej zmiennych do węzłów grafu. Funkcja add_state_random() służy do uzupełnienia grafu o zmienną określającą początkową liczbę węzłów posiadających informację.
graph_init()
Funkcja graph_init() odpowiada za utworzenie przykładowego losowego grafu do przeprowadzenia symulacji. Generowany jest domyślnie graf Wattsa Strogatza, odwzorowujący sieci małego świata. Pierwsze trzy argumenty są bezpośrednio przekazywane do funkcji watts_strogatz_graph() z biblioteki NetworkX. Funkcja posiada łącznie sześć argumentów:
1) n – typ integer, oznacza liczbę węzłów w grafie,
2) k – typ integer, stanowi o liczbie sąsiadów dla każdego węzła przed losowym przepisywaniem krawędzi,
3) rewire_prob – typ float, określa prawdopodobieństwo przepisania krawędzi w losowe miejsce w grafie,
4) initiation_perc – typ float, oznacza odsetek węzłów wybieranych losowo, które dysponują informacją na samym początku symulacji,
5) show_attr – typ bool, odpowiada za wyświetlenie wag i atrybutów węzłów po utworzeniu grafu,
6) draw_graph – typ bool, tworzy wykres grafu.
Funkcja zwraca poniższe obiekty:
1) G – graf, który jest obiektem klasy grafu NetworkX, oraz
2) pos – zmienna typu ndarray, która przechowuje pozycje węzłów grafu niezbędne do wizualizacji.
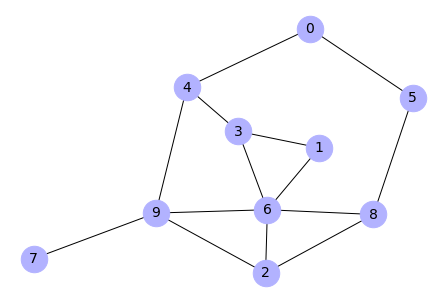
Poniższym poleceniem tworzony jest graf losowy Wattsa-Strogatza o dziesięciu węzłach (n = 10), o średniej ilości połączeń między wezłami równej 5 (k = 5), prawdopodobieństwie przełączenia danego wiązania równym 0.1 (rewire_prob = 0.1), początkowej liczbie agentów dysponujących informacją równej 10% populacji (initiation_perc = 0.1). Dodatkowo funkcja wyświetla atrybuty węzłów wraz z wagami (show_attr = True), oraz tworzy graf (draw_graph = True).
Input:
G, pos = dp.graph_init(n = 10,
k= 5,
rewire_prob = 0.1,
initiation_perc = 0.1,
show_attr = True,
draw_graph = True)
Output:
Node attributes:
0 {'state': 'unaware', 'receptiveness': 1.0, 'extraversion': 0.582378, 'engagement': 1e-06}
1 {'state': 'unaware', 'receptiveness': 0.800181, 'extraversion': 0.329359, 'engagement': 0.028751}
2 {'state': 'unaware', 'receptiveness': 0.385395, 'extraversion': 0.226138, 'engagement': 0.623113}
3 {'state': 'aware', 'receptiveness': 0.368734, 'extraversion': 0.14079, 'engagement': 0.398072}
4 {'state': 'unaware', 'receptiveness': 0.306774, 'extraversion': 0.244501, 'engagement': 0.023409}
5 {'state': 'unaware', 'receptiveness': 0.275711, 'extraversion': 0.879711, 'engagement': 0.275111}
6 {'state': 'unaware', 'receptiveness': 0.578056, 'extraversion': 0.308108, 'engagement': 0.786855}
7 {'state': 'unaware', 'receptiveness': 0.844748, 'extraversion': 1e-06, 'engagement': 0.838986}
8 {'state': 'unaware', 'receptiveness': 0.702168, 'extraversion': 0.578449, 'engagement': 0.294877}
9 {'state': 'unaware', 'receptiveness': 0.807228, 'extraversion': 0.808263, 'engagement': 0.866605}
Wages:
0 [1.e-06]
1 [0.012265]
2 [0.020133]
3 [0.046409]
4 [0.053141]
5 [0.089238]
6 [0.094578]
7 [0.095085]
8 [0.23072]
9 [0.245545]
10 [0.271505]
11 [0.29906]
12 [0.310618]
13 [0.38126]
14 [0.397444]
15 [0.467515]
16 [0.512404]
17 [0.591285]
18 [0.641824]
19 [1.]
W ramach działania funkcji automatycznie generowane są dla grafu losowe wagi, losowane z rozkładu wykładniczego i oznaczają prawdopodobieństwa kontaktu między poszczególnymi agentami. Przypisywane są one do krawędzi grafu, czyli odpowiadają relacjom między węzłami. Dodatkowo generowane są cztery atrybuty przypisane do węzłów. Są to kolejno receptiveness – receptywność, extraversion – ekstrawersja, engagement – zaangażowanie, state – stan węzła. Poziom receptywności jest losowany z rozkładu normalnego i oznacza zdolność agenta do odbierania bodźców ze środowiska. Poziom ekstrawersji jest również generowany z rozkładu normalnego i odpowiada poziomowi ekstrawersji agenta w psychologicznej koncepcji klasyfikacji osobowości Carla G. Junga. Zaangażowanie oznacza poziom zaangażowania aktora w dany temat, na ile jest on związany swoim doświadczeniem z tematem w którego zakresie leży informacja. Zaangażowanie jest losowane z rozkładu wykładniczego. Wszystkie trzy atrybuty ilościowe są skalowane do przedziału (0,1] w celu ułatwienia przeprowadzenia symulacji, która odwołuje się później do tych wartości. Stan węzła jest atrybutem dwupoziomowym i przyjmuje wartość aware – świadomy i unaware – nieświadomy w kontekście posiadania danej informacji. Powyższe cechy służą później do dobliczania szansy na przeniesienie się informacji między poszczególnymi węzłami. Szansa ta jest obliczana dla każdej relacji między węzłami w jądrze symulacji nazwanym “WERE” zaimplementowanym w module simulate.py. Nazwa “WERE” to akronim od nazw parametrów biorących udział w obliczeniach. Pakiet umożliwia wykorzystanie dowolnego własnego równania, zamieniając jądro “WERE” na własną funkcję.
draw_graph()
Funkcja draw_graph() odpowiedzialna jest za wizualizację grafu. Może być wywoływana z poziomu funkcji graph_init(), z argumentem draw_graph == True. Funkcja draw_graph() odwołuje się do biblioteki NetworkX z funkcjami draw_networkx_nodes(), draw_networkx_labels(), draw_networkx_edges(), które z kolei bazują na bibliotece Matplotlib. Funkcja przyjmuje pięć argumentów:
1) G – obiekt grafowy biblioteki NetworkX, ze zdefiniowaną zmienną state przypisaną do węzłów,
2) pos – zmienna typu ndarray, która przechowuje pozycje węzłów grafu niezbędne do wizualizacji,
3) aware_color – zmienna typu string, która określa kolor węzłów świadomych danej informacji,
przyjmuja wartości określające kolor w zapisie Hex
4) not_aware_color – zmienna typu string, która określa kolor węzłów nieświadomych danej informacji,
5) legend – zmienna typu bool, która odpowiada za dodanie legendy do wykresu.
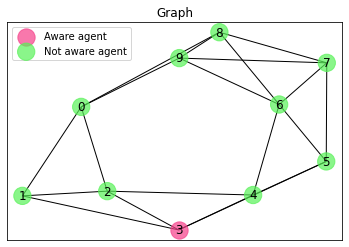
Aby wykorzystać tę funkcję, niezbędny jest atrybut state przypisany do węzłów grafu, z wartościami aware i unaware określającymi, które węzły są w posiadaniu informacji. Funkcja wyświetla wykres grafu.
Input:
dp.draw_graph(G, # graph
pos, # position of nodes
aware_color = '#f63f89',
not_aware_color = '#58f258',
legend = True)
Output:

graph_stats()
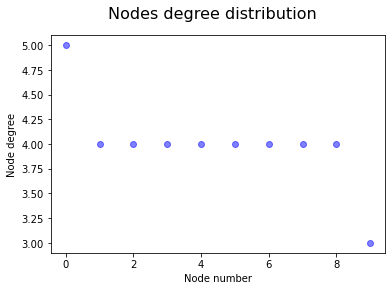
Funkcja graph_stats() służy do obliczania podstawowych statystyk grafu. Funkcja przyjmuje pięć argumentów:
1) G – obiekt klasy grafu biblioteki NetworkX,
2) pos – zmienna typu ndarray, która przechowuje pozycje węzłów grafu niezbędne do wizualizacji,
3) show_attr – typ bool, umożliwia wyświetlenie wag i atrybutów węzłów danego grafu,
4) draw_degree – typ bool, określenie wartości jako True odpowiada za utworzenie wykresu rozkładu stopni węzłów grafu,
5) draw_graph – typ bool, umożliwia utworzenie wykresu grafu.
Funkcja zwraca obiekt dict_stat – typu
słownikowego, zawierający podstawowe statystyki grafu, takie jak liczbę węzłów sieci, liczbę krawędzi,
wagi połączeń między węzłami, atrybuty węzłów.
Input:
dp.graph_stats(G,
pos,
show_attr = False,
draw_degree = True,
draw_graph = False)
Output:
General information:
nodes : 10
edges : 20
mean node degree : 4
average clustering coefficient : 0.4833
transitivity : 0.4918
add_feature()
Funkcja add_feature() odpowiada za dodanie atrybutów do grafu z opcjonalnym skalowaniem tych atrybutów do przedziału (0,1]. Wywołując funkcję graph_init() zmienne są przyporządkowywane do węzłów automatycznie, natomiast funkcja add_feature() daje większą kontrolę nad dodawaną zmienną. Funkcja przyjmuje osiem argumentów:
1) G – obiekt klasy grafu biblioteki NetworkX,
2) pos – zmienna typu ndarray, która przechowuje
pozycje węzłów grafu niezbędne do wizualizacji,
3) feature – zmienna typu ndarray, będąca atrybutem przypisywanym do węzłów grafu,
4) feature_type – zmienna typu string, mająca poziomy: “weights”, “receptiveness”,
“extraversion”, “engagement”, “state”, lub inne dowolne nazwy,
5) scaling – typ bool, argument opcjonalny,
umożliwia skalowanie dodanej zmiennej do przedziału (0,1],
6) decimals – typ integer, argument opcjonalny,
odpowiada za liczbę cyfr po przecinku przy zaokrąglaniu,
7) show_attr – typ bool, umożliwia wyświetlenie wag
i atrybutów węzłów danego grafu,
8) draw_graph – typ bool, argument opcjonalny,
odpowiada za utworzenie wykresu grafu.
Funkcja zwraca zmienną G – graf, który jest obiektem klasy grafu NetworkX.
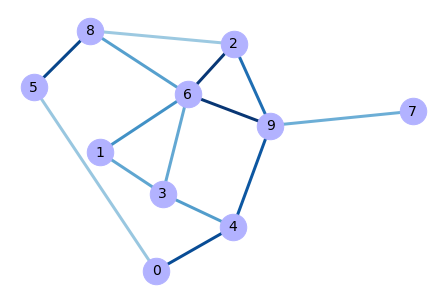
Input:
import networkx as nx
# Create basic watts-strogatz graph
G_02 = nx.watts_strogatz_graph(n = 10, k = 6, p = 0.3, seed=None)
# Compute a position of graph elements
pos = nx.spring_layout(G_02)
import numpy as np
# Create example weights
weights = np.round(np.random.exponential(scale = 0.1,
size = G_02.number_of_edges()), 6).reshape(G_02.number_of_edges(),1)
# Add feature
G_02 = dp.add_feature(G_02,
pos,
feature = weights,
feature_type = "weights",
scaling = True,
decimals = 6,
show_attr = True, # show node weights and attributes
draw_graph = False)
Output:
Nodes' attributes:
0 {}
1 {}
2 {}
3 {}
4 {}
5 {}
6 {}
7 {}
8 {}
9 {}
Sorted weights:
0 1e-06
1 0.010339
2 0.01119
3 0.038441
4 0.049669
5 0.051154
6 0.068371
7 0.081067
8 0.082919
9 0.086678
10 0.110474
11 0.121775
12 0.12913
13 0.132083
14 0.152151
15 0.164671
16 0.182431
17 0.187515
18 0.245855
19 0.268703
20 0.339081
21 0.407199
22 0.545156
23 0.560313
24 0.654668
25 0.683216
26 0.713856
27 0.770032
28 0.98711
29 1.0
Input:
# Create example engagement
engagement = np.round(np.random.exponential(scale = 0.1,
size = G_02.number_of_edges()), 6).reshape(G_02.number_of_edges(),1)
# Add feature
G_02 = dp.add_feature(G_02,
pos,
feature = engagement,
feature_type = "engagement",
scaling = True,
show_attr = True, # show node weights and attributes
draw_graph = False)
Output:
Nodes' attributes:
0 {'engagement': 0.28056}
1 {'engagement': 0.196385}
2 {'engagement': 0.001351}
3 {'engagement': 1.0}
4 {'engagement': 0.187343}
5 {'engagement': 0.004473}
6 {'engagement': 0.305583}
7 {'engagement': 0.10013}
8 {'engagement': 0.113332}
9 {'engagement': 0.031052}
Sorted weights:
0 1e-06
1 0.010339
2 0.01119
3 0.038441
4 0.049669
5 0.051154
6 0.068371
7 0.081067
8 0.082919
9 0.086678
10 0.110474
11 0.121775
12 0.12913
13 0.132083
14 0.152151
15 0.164671
16 0.182431
17 0.187515
18 0.245855
19 0.268703
20 0.339081
21 0.407199
22 0.545156
23 0.560313
24 0.654668
25 0.683216
26 0.713856
27 0.770032
28 0.98711
29 1.0
add_state_random()
Funkcja add_state_random() odpowiada za wygenerowanie zmiennej określającej czy dany aktor w sieci jest świadomy określonej informacji. Funkcja przyjmuje pięć parametrów:
1) G – obiekt grafowy biblioteki NetworkX,
2) pos – typ ndarray, przechowuje
pozycje węzłów grafu niezbędne do wizualizacji,
3) initiation_perc – typ float, określa odsetek węzłów
dysponujących początkowo informacją,
4) show_attr – typ bool, odpowiada za wyświetlenie wag
i atrybutów węzłów danego grafu,
5) draw_graph – typ bool, opcjonalna, umożliwia utworzenie
wykresu grafu.
Funkcja zwraca zmienną G – obiekt klasy grafu NetworkX.
Input:
# add state
dp.add_state_random(G_02,
pos,
initiation_perc = 0.2,
show_attr = True,
draw_graph = True)
Output:
Node attributes:
0 {'engagement': 0.28056, 'state': 'aware'}
1 {'engagement': 0.196385, 'state': 'unaware'}
2 {'engagement': 0.001351, 'state': 'aware'}
3 {'engagement': 1.0, 'state': 'unaware'}
4 {'engagement': 0.187343, 'state': 'unaware'}
5 {'engagement': 0.004473, 'state': 'unaware'}
6 {'engagement': 0.305583, 'state': 'unaware'}
7 {'engagement': 0.10013, 'state': 'unaware'}
8 {'engagement': 0.113332, 'state': 'unaware'}
9 {'engagement': 0.031052, 'state': 'unaware'}
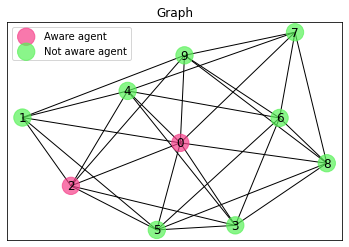
2. Moduł symulacyjny
Moduł simulate.py służy do przeprowadzenia symulacji rozchodzenia się informacji w sieciach. Składa się z funkcji simulation_step(), simulation() i simulation_sequence(). W module simulate.py istnieje możliwość wykorzystania funkcji simulation_step() do generowania pojedynczych kroków symulacji. Na każdym kroku symulacji można tworzyć wykres bieżącej struktury sieci. Funkcja simulation() do symulacji wielokrokowych wywołuje funkcję simulation_step() i dodaje nowe funkcjonalności udostępniając parametry funkcji simulation_step() oraz dodatkowy parametr liczby przeprowadzonych symulacji. Analogicznie działa funkcja simulation_sequence() do sekwencji symulacji wielokrokowych, która opakowuje funkcję simulation() i udostępnia również dodatkowe parametry. Taka struktura umożliwia wykorzystanie funkcji simulation_step() jako podstawy symulacji i dopisywanie własnych dodatkowych elementów kodu w razie potrzeby. W funkcji simulation_sequence() zaimplementowano metodę Monte Carlo. Wykorzystano ją do obliczania zdolności propagacji informacji zadanego zestawu węzłów. Element mechanizmu losowego przeszukiwania zaszyty jest w funkcji simulation_step(), w której proces symulacyjny jest oparty o zmienne losowe, wyrażające prawdopodobieństwa kontaktu między węzłami w sieci.
simulation_step()
Funkcja simulation_step() wykonuje jeden krok symulacji i nadpisuje zmiany parametrów w grafie. Przyjmuje osiem argumentów:
1) G – obiekt klasy grafu biblioteki NetworkX,
2) pos – przyjmuje zmienną typu ndarray, która przechowuje
pozycje węzłów grafu niezbędne do wizualizacji.
3) kernel – typ string, posiada poziomy “weights”, “WERE” i “custom”.
Parametr określony jako “weights” aplikuje do symulacji jądro, które
podczas rozprzestrzeniania się informacji bierze pod uwagę tylko wagi
na krawędziach między węzłami. Po ustawieniu parametru jako “WERE” – do
obliczania prawdopodobieństwa przekazania informacji między węzłami
jest aplikowane równanie oparte na parametrach weights-extraversion-receptiveness-engagement
przyporządkowanych do węzłów sieci. Aby uruchomić symulację z tym jądrem,
graf powinien mieć zdefiniowane powyższe zmienne, przypisane
do wierzchołków grafu. Natomiast z wartością “custom” – prawdopodobieństwo
rozchodzenia się informacji jest obliczane z własną zdefiniowaną funkcją,
4) Engagement_enforcement – typ float, liczba podana jako argument
jest mnożnikiem parametru engagement dla poszczególnych węzłów,
które są już świadome, lub zapomniały informację po danej iteracji,
5) custom_kernel – dowolna funkcja obliczająca prawdopodobieństwo
przekazania informacji w każdym kroku symulacji dla każdego węzła.
Funkcja może operować na zmiennych przypisanych do wierzchołków grafu,
6) WERE_multiplier – typ float, argument opcjonalny, odpowiada
za regulację wartości obliczanej przez jądro “WERE”,
7) oblivion – typ bool, argument opcjonalny, odpowiada
za zapominanie agentów o informacji.
8) draw – typ bool, argument opcjonalny, umożliwia utworzenie
wykresu grafu.
Funkcja zwraca zmienną G – graf, który jest obiektem klasy grafu NetworkX. Poniżej przeprowadzone są dwa kroki symulacji na grafie zaprezentowanym powyżej, podczas omawiania funkcji add_state_random() .
Input:
# Copy graph
import copy
G_03 = copy.deepcopy(G_02)
# Perform one simulation step
G_03 = dp.simulation_step(G_03,
pos,
kernel = 'weights',
custom_kernel = None,
WERE_multiplier = 10,
oblivion = False,
engagement_enforcement = 1.01,
draw = True,
show_attr = False)
Output:
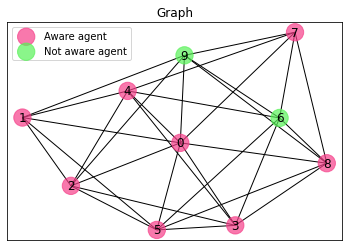
Input:
# Perform one simulation step
G_03 = dp.simulation_step(G_03,
pos,
kernel = 'weights',
custom_kernel = None,
WERE_multiplier = 10,
oblivion = False,
engagement_enforcement = 1.01,
draw = True,
show_attr = False)
Output:
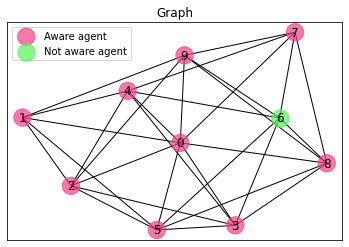
simulation()
Funkcja simulation() wykonuje zadaną liczbę kroków symulacji i zwraca graf z nadpisanymi parametrami. Funkcja przyjmuje dziewięć argumentów – osiem argumentów zapożyczonych z funkcji simulation_step(), oraz dodatkowy argument n o typie integer, określający liczbę kroków w symulacji. Funkcja zwraca następujące zmienne:
1) G – graf, który jest obiektem klasy grafu NetworkX,
2) graph_list, zmienną typu lista przechowującą informacje
o stanie węzłów we wszystkich krokach sytuacji.
Lista składa się wewnętrznych list.
Każda wewnętrzna lista odnosi się do odpowiadającego jej kroku w symulacji.
3) avg_aware_inc_per_step – lista przechowująca średni
przyrost świadomych agentów na jeden krok symulacji.
Input:
# Copy graph
G_03 = copy.deepcopy(G_02)
# Simulation
graph_list, avg_aware_inc_per_step \
= dp.simulation(G_03,
pos,
n = 2,
kernel = 'weights',
custom_kernel = None,
WERE_multiplier = 10,
oblivion = False,
engagement_enforcement = 1.01,
draw = False,
show_attr = False)
print(avg_aware_inc_per_step)
import pprint
pprint.pprint(graph_list)
Output:
3.5
[[(0, {'engagement': 0.28056, 'state': 'aware'}),
(1, {'engagement': 0.196385, 'state': 'unaware'}),
(2, {'engagement': 0.001351, 'state': 'aware'}),
(3, {'engagement': 1.0, 'state': 'unaware'}),
(4, {'engagement': 0.187343, 'state': 'unaware'}),
(5, {'engagement': 0.004473, 'state': 'unaware'}),
(6, {'engagement': 0.305583, 'state': 'unaware'}),
(7, {'engagement': 0.10013, 'state': 'unaware'}),
(8, {'engagement': 0.113332, 'state': 'unaware'}),
(9, {'engagement': 0.031052, 'state': 'unaware'})],
[(0, {'engagement': 0.291953, 'state': 'aware'}),
(1, {'engagement': 0.202335, 'state': 'aware'}),
(2, {'engagement': 0.001407, 'state': 'aware'}),
(3, {'engagement': 1.0, 'state': 'unaware'}),
(4, {'engagement': 0.187343, 'state': 'unaware'}),
(5, {'engagement': 0.004518, 'state': 'aware'}),
(6, {'engagement': 0.305583, 'state': 'unaware'}),
(7, {'engagement': 0.10013, 'state': 'unaware'}),
(8, {'engagement': 0.113332, 'state': 'unaware'}),
(9, {'engagement': 0.031363, 'state': 'aware'})],
[(0, {'engagement': 0.309914, 'state': 'aware'}),
(1, {'engagement': 0.210551, 'state': 'aware'}),
(2, {'engagement': 0.001478, 'state': 'aware'}),
(3, {'engagement': 1.040604, 'state': 'aware'}),
(4, {'engagement': 0.187343, 'state': 'unaware'}),
(5, {'engagement': 0.004796, 'state': 'aware'}),
(6, {'engagement': 0.314842, 'state': 'aware'}),
(7, {'engagement': 0.10013, 'state': 'aware'}),
(8, {'engagement': 0.11561, 'state': 'aware'}),
(9, {'engagement': 0.032963, 'state': 'aware'})]]
simulation_sequence()
Funkcja simulation_sequence() wykonuje sekwencję symulacji. Przekazuje ona argumenty do funkcji simulation(), oraz pobiera dodatkowy argument sequence_len o typie integer, określający liczbę symulacji w sekwencji. Funkcja zwraca zmienną avg_aware_inc typu float, która zawiera średni przyrost świadomych agentów na jeden krok dla wszystkich symulacji. Funkcja nie zwraca zmodyfikowanego grafu.
Input:
avg_aware_inc_per_step = \
dp.simulation_sequence(G_03, # networkX graph object
#pos, # p
n = 2, # number of steps in simulation
sequence_len = 1000, # sequence of simulations
kernel = 'weights', # kernel type
custom_kernel = None, # custom kernel function
WERE_multiplier = 10,
oblivion = False, # information oblivion feature
engagement_enforcement = 1.01,
show_attr = False)
Output:
3.6325
3. Moduł optymalizacyjny
Celem działania funkcji z modułu optymalizacyjnego jest wyznaczenie n węzłów sieci, które najszybciej rozpropagują informację. W skład modułu wchodzi funkcja optimize_centrality() pozwalająca na obliczanie zestawu n węzłów cechujących się największym poziomem bliskości (ang. closeness) w sieci w stosunku do innych węzłów. Drugą funkcją jest optimize_rs(), która poszukuje najlepszych wierzchołków symulacyjnie za pomocą metody przeszukiwania losowego.
optimize_centrality()
Funkcja optimize_centrality() przyjmuje następujące argumenty:
1) G – graf, który jest obiektem klasy grafu NetworkX,
2) number_of_nodes – typ integer, oznacza liczbę węzłów do wytypowania.
Kolejna dwa argumenty przekazywane są do funkcji wyliczającej
miarę bliskości węzłów z biblioteki NetworkX:
3) distance – typ string, parametr opcjonalny,
wskazuje na nazwę atrybutu krawędzi, która ma zostać użyta do optymalizacji,
4) wf_improved – typ bool, argument przyjmuje wartość logiczną
określającą użycie ulepszonej wersji algorytmu wyliczającego bliskość.
Funkcja zwraca obiekt n_best_nodes będący listą krotek,
gdzie każda krotka zawiera numer węzła oraz jego miarę centralności.
Do aproksymacji centralności węzła została zastosowana funkcja
obliczająca bliskość (ang. closeness) między wierzchołkami grafu.
Input:
# Copy graph
G_03 = copy.deepcopy(G_02)
# Run function with weights as distances
dp.optimize_centrality(G_03,
distance = 'weight',
wf_improved = False,
number_of_nodes = 4)
Output:
[(0, 13.02914620004951),
(4, 13.02914620004951),
(1, 13.029033028598729),
(2, 9.811242593874512)]
optimize_rs()
Funkcja optimize_rs() przyjmuje osiem parametrów, które przekazuje do funkcji simulation_sequence:
1) G – obiekt klasy grafu biblioteki NetworkX,
2) n – typ integer; odpowiedzialny za liczbę kroków w symulacji,
3) sequence_len – parametr typu integer określający
liczbę symulacji w jednej sekwencji,
4) kernel – typ string, posiada poziomy “weights”, “WERE” i “custom”,
5) custom_kernel – dowolna funkcja obliczająca prawdopodobieństwo
przekazania informacji w każdym kroku symulacji dla każdego węzła.
Funkcja może operować na zmiennych przypisanych do wierzchołków grafu,
6) WERE_multiplier – typ float, argument opcjonalny, odpowiada
za regulację wartości obliczanej przez jądro “WERE”,
7) oblivion – typ bool, argument opcjonalny, odpowiada
za zapominanie agentów o informacji.
8) Engagement_enforcement – typ float, liczba podana jako argument
jest mnożnikiem parametru engagement dla poszczególnych węzłów,
oraz parametry:
9) number_of_nodes – typ integer, określa liczbę węzłów do wytypowania,
10) number_of_iter – typ integer, wyznacza liczbę iteracji w optymalizacji random search,
11) log_info_interval – typ integer, parametr opcjonalny,
odpowiada za interwał iteracji pomiędzy kolejnymi wyświetleniami
w konsoli informacji o postępie optymalizacji. Wartość ustawiona
jako None ukrywa wyświetlanie informacji.
Input:
solution = dp.optimize_rs(G_03,
number_of_nodes = 2, # number of nodes to seed
number_of_iter = 1000, # number of iterations
log_info_interval = 1, # interval of information log
n = 3, # number of simulation steps simulation
sequence_len = 100, # number of simulations in one seq
kernel = 'weights', # kernel type
custom_kernel = None, # custom kernel function
WERE_multiplier = 10,
oblivion = False, # information oblivion feature
engagement_enforcement = 1.00
)
Output:
2 Iterations passed with best solution: 2.62 in 1.19 seconds.
3 Iterations passed with best solution: 2.62 in 1.59 seconds.
4 Iterations passed with best solution: 2.62 in 1.94 seconds.
5 Iterations passed with best solution: 2.62 in 2.38 seconds.
6 Iterations passed with best solution: 2.62 in 2.77 seconds.
7 Iterations passed with best solution: 2.6567 in 3.27 seconds.
8 Iterations passed with best solution: 2.6567 in 3.6 seconds.
9 Iterations passed with best solution: 2.6567 in 4.02 seconds.
10 Iterations passed with best solution: 2.6567 in 4.44 seconds.
.
.
995 Iterations passed with best solution: 2.6667 in 462.58 seconds.
996 Iterations passed with best solution: 2.6667 in 463.31 seconds.
997 Iterations passed with best solution: 2.6667 in 463.79 seconds.
998 Iterations passed with best solution: 2.6667 in 464.45 seconds.
999 Iterations passed with best solution: 2.6667 in 465.16 seconds.
Best aware agents increment per simulation step: 2.6666666666666634
Set of initial aware nodes: [4, 1]
4. Moduł modelująco-wyjaśniający
Celem działania modułu modelująco-wyjaśniającego jest rozpoznanie, które atrybuty węzłów sieci implikują zdolność tych węzłów do efektywnego rozprowadzania informacji. W tym celu niezbędne jest utworzenie zbioru danych, w którym określimy jak efektywnie poszczególne węzły rozprowadzają informację. Może to odbyć się na drodze przeprowadzania symulacji i sprawdzania zdolności dla każdego węzła (lub próby tych węzłów), lub na drodze obliczania miar centralności. Kiedy przeprowadzone zostaną wymagane obliczenia, utworzona zostanie nowa zmienna, która będzie zmienną wyjaśnianą w modelu. Natomiast zmiennymi wyjaśniającymi będą pozostałe atrybuty przyporządkowane do węzłów sieci. Po utworzeniu modelu matematycznego na takim zbiorze danych należy wykorzystać jedną z technik analitycznych umożliwiających rozpoznanie istotności, czy też wkładu poszczególnych zmiennych w wyjaśnianie zmienności wariancji zmiennej zależnej. W module zastosowano funkcję XGBRegressor() z biblioteki XGBoost, która modeluje dane. Następnie na modelu stosowana jest metoda feature_importances_() obliczająca istotność zmiennych w modelu. Po wyliczanej istotności można ocenić, które zmienne dotyczące węzłów mogą być wskaźnikami zdolności do szybkiej propagacji informacji po sieci. Tego typu zabieg dostarcza informacji jakie cechy węzłów są istotne z biznesowego punktu widzenia. Przekładając to na przykładowy problem biznesowy – dowiemy się jakimi kryteriami powinien posłużyć się decydent, aby wybrać influencera do swojej kampanii marketingowej.
Moduł zawiera dwie funkcje: nodes_score_simulation() służącą do obliczenia miary nośności informacji dla każdego węzła, oraz feature_importance() odpowiadającą za obliczanie związku innych atrybutów węzłów z metryką wyliczoną przez nodes_score_simulation().
nodes_score_simulation()
Funkcja nodes_score_simulation() odpowiada za obliczenie średniego przyrostu świadomych agentów w sieci na jeden krok symulacji dla każdego węzła jako początkowego seedera. Funkcja przyjmuje dziewięć parametrów:
1) log_info_interval – typ integer, parametr opcjonalny,
odpowiada za interwał iteracji pomiędzy kolejnymi wyświetleniami
w konsoli informacji o postępie w obliczaniu miary centralności
dla węzłów. Ustawiona wartość jako None ukrywa wyświetlanie
informacji.
Osiem pozostałych parametrów przekazywanych jest
kolejno do funkcji simulation_sequence(), oraz do bardziej
zagnieżdżonych funkcji simulation() i simulation_step, i są to kolejno:
2) G – graf biblioteki NetworkX,
3) sequence_len – parametr typu integer określający
liczbę symulacji w jednej sekwencji,
4) n – typ integer; odpowiedzialny za liczbę kroków w symulacji,
5) kernel – typ string, nazwa jądra symulacji,
posiada poziomy “weights”, “WERE” i “custom”,
6) engagement_enforcement – typ float, liczba podana jako argument
jest mnożnikiem parametru engagement dla poszczególnych węzłów, którę są
już świadome, lub zapomniały informację po danej iteracji,
7) custom_kernel – dowolna funkcja obliczająca prawdopodobieństwo
przekazania informacji w każdym kroku symulacji dla każdego węzła,
8) WERE_multiplier – typ float, argument opcjonalny, odpowiada
za regulację wartości obliczanej przez jądro WERE,
9) oblivion – typ bool; opcjonalny; umożliwia
włączenie opcji zapominania informacji przez agentów.
Funkcja zwraca obiekt list_solution będący listą z obliczonymi miarami szybkości propagacji informacji dla pojedynczych węzłów. Każda wartość to średni przyrost świadomych węzłów w sieci na jeden krok symulacji.
Input:
solution = dp.nodes_score_simulation(G_03,
log_info_interval = 1, # interval of information log
n = 3, # number of simulation steps simulation
sequence_len = 100, # number of simulations in one seq
kernel = 'weights', # kernel type
custom_kernel = None, # custom kernel function
WERE_multiplier = 10,
oblivion = False, # information oblivion feature
engagement_enforcement = 1.00
)
Output:
1 Iterations passed in 0.89 seconds.
2 Iterations passed in 1.28 seconds.
3 Iterations passed in 1.66 seconds.
4 Iterations passed in 2.0 seconds.
5 Iterations passed in 2.35 seconds.
6 Iterations passed in 2.71 seconds.
7 Iterations passed in 3.0 seconds.
8 Iterations passed in 3.31 seconds.
9 Iterations passed in 3.54 seconds.
List of solutions: [2.7766666666666664, 2.783333333333333,
2.9166666666666656, 2.833333333333333, 2.733333333333333,
2.7666666666666657, 2.7200000000000006, 2.453333333333333,
2.6166666666666663, 2.1999999999999993]
feature_importance()
Funkcja feature_importance() służy do obliczania związku
pomiędzy zmienną przechowującą miary węzłów, a innymi zmiennym przypisanymi do węzłów.
Funkcja przyjmuje 11 argumentów, z czego 9 argumentów jest identycznych
z oczekiwanymi w funkcji nodes_score_simulation()
i są one do niej przekazywane. Pozostałe dwa argumenty to:
1) X – typ ndarray, przyjmuje dwuwymiarową tablicę ze zmiennymi, których
związek będzie sprawdzany ze zmienną zwróconą przez funkcję nodes_score_simulation(),
2) show – typ bool, odpowiada za wyświetlenie wyników w konsoli po zakończeniu działania funkcji.
Poniżej do zmiennej Y przypisano obliczone miary nośności informacji dla węzłów grafu G_03 wykorzystywanego w przykładach powyżej. Utworzono również 3 sztuczne zmienne skorelowane ze zmienną X w celu przetestowania funkcji feature_importance().
Input:
import numpy as np
import math
from scipy.linalg import toeplitz, cholesky
from statsmodels.stats.moment_helpers import cov2corr
#=======================================#
# Create theoretical correlation matrix #
#=======================================#
p = 4 # total number of variables
h = 2/p
# create vector as a base for our next correlation matrix
v = np.linspace(1,-1+h,p)
# Create theoretical correlation matrix
R = cov2corr(toeplitz(v))
#===========================#
# create the first variable # (our "given variable")
# ==========================#
Y = solution
#=================================#
# generate p-1 correlated randoms #
#=================================#
# Generate 4 random variables
X_01 = np.random.randn(len(G_03),p)
# In 1 column insert our existing variable
X_01[:,0] = Y
# Cholesky decomposition (create some kind of matrix)
C = cholesky(R)
# Matrix product of two arrays. (iloczyn macierzowy)
X = np.matmul(X_01,C)
# set 6 digit decimal
np.set_printoptions(precision=6)
np.set_printoptions(suppress=True)
# remove 1st column (target variable)
X = np.delete(X, 0, 1)
FI = dp.feature_importance(
G_03, # NetworkX graph
X, # Nodes attributes
show = True,
log_info_interval = 10, # interval of information log
n = 3, # number of simulation steps simulation
sequence_len = 20, # number of simulations in one seq
kernel = 'weights', # kernel type
custom_kernel = None, # custom kernel function
WERE_multiplier = 10,
oblivion = False, # information oblivion feature
engagement_enforcement = 1.00
)
Output:
List of solutions: [2.95, 2.9166666666666665, 2.8333333333333335,
2.8166666666666664, 2.7333333333333334, 2.833333333333333,
2.7666666666666666, 2.65, 2.6666666666666665, 1.85]
Feature importances:
Variable 1 : 0.8862867
Variable 2 : 0.03555947
Variable 3 : 0.07815386